Qué es Latin-SEQ:
Latin-SEQ es un proyecto de investigación cuyo objetivo principal es proporcionar diagnóstico genético a personas afectadas por enfermedades neuromusculares (ENM) hereditarias residentes en países de Latinoamérica (LATAM). Con este fin, utilizamos una serie de técnicas genéticas, fundamentalmente «Next Generation Sequencing» (NGS) pero también secuenciación tradicional usando Sanger y técnicas de MLPA. Se trata de un estudio internacional y multicéntrico en el que participan más de 60 centros hospitalarios en 18 países, coordinado por el John Walton Muscular Dystrophy Research Center (JWMDRC) en la Universidad de Newcastle, Reino Unido.
Nuestro compromiso es acelerar el proceso de diagnóstico genético de estos pacientes y expandir el conocimiento sobre las causas y la prevalencia de las enfermedades neuromusculares en diferentes poblaciones LATAM. Obtener un diagnóstico precoz ofrece a los pacientes una multitud de beneficios: acceso a un plan de seguimiento y terapia personalizada, expectativas claras sobre la progresión de su enfermedad, posibilidad de obtener consejo genético para los familiares, posibilidad de inclusión en registros o asociaciones de pacientes y, participación en ensayos clínicos si los hubiese.
Latin-SEQ fomenta la creación de una sólida red de unidades hospitalarias y centros clínicos con experiencia en enfermedades neuromusculares que siguen a pacientes adultos y pediátricos abarcando toda LATAM.
Latin-SEQ también incluye espacios virtuales para la formación especializada, el debate conjunto de casos clínicos y la colaboración en proyectos de investigación reuniendo a neurólogos, neuropediatras, neurofisiólogos, genetistas, fisioterapeutas y otros profesionales de la salud que estén involucrados en el diagnóstico y seguimiento de pacientes con enfermedades neuromusculares.
Objetivos
Qué es secuenciado de exomas:
La secuenciación es una técnica molecular que permite analizar el ADN de una persona para “leer” su código genético. Anteriormente, se utilizaban métodos como la secuenciación de Sanger, que, aunque preciso, era más lento, ya que solo permite secuenciar una única región del ADN (gen) a la vez.
Hoy en día, utilizamos técnicas más avanzadas como la Secuenciación de Nueva Generación (NGS). Esta nueva tecnología es más rápida y permite secuenciar grandes regiones de ADN simultáneamente y en poco tiempo, lo cual facilita y acelera el diagnóstico genético. En el proyecto Latin-SEQ, usamos Secuenciación de Exoma Completo (WES por sus siglas en inglés), a modo de obtener información de todas las regiones codificantes, es decir aquellas que contienen las instrucciones para producir todas las proteínas. Al comparar los exomas de un individuo afecto, con una secuencia de referencia, podemos identificar variantes genéticas que puedan ser la causa de estas enfermedades neuromusculares. Gracias a esta tecnología, podemos ofrecer un diagnóstico más rápido y preciso, mejorando así la atención y la calidad de vida de los pacientes.
Criterios de inclusión y exclusión:
Pueden ser incluidos en el proyecto Latin-SEQ todos aquellos pacientes con sospecha de enfermedad neuromuscular de origen genético aun sin diagnóstico. No hay restricción de sexo ni de edad (pacientes adultos como pediátricos), ni tampoco respecto al tipo de enfermedad neuromuscular que presenten (tanto músculo y placa como nervio); solo quedaran excluidas las enfermedades adquiridas o aquellas que no puedan ser diagnosticadas mediante el análisis de exoma (DM1/2 y FSHD). El estudio priorizará el análisis de familias con múltiples miembros afectados y pacientes de minorías étnicas.
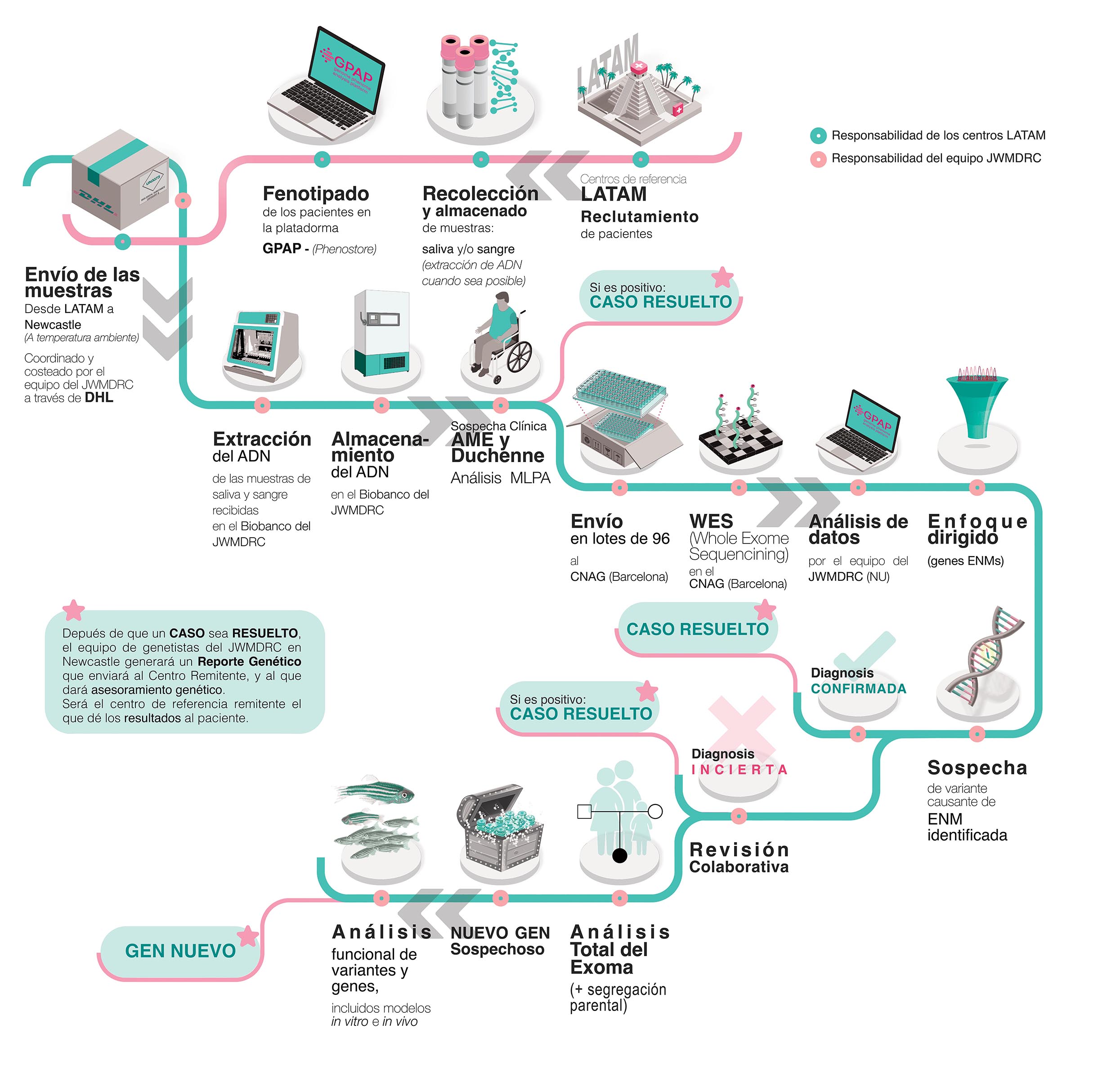
Reclutamiento, toma de muestras y fenotipado:
Los profesionales sanitarios de cada centro participante seleccionarán sus pacientes y se asegurarán de que los participantes firmen el consentimiento informado. Además, se encargarán de tomar las muestras biológicas (ADN, sangre, saliva o gotas de sangre seca en función de sus posibilidades). Estas serán enviadas y almacenadas en el biobanco del “JWMDRC Biobank” en Newcastle, hasta su análisis. Además, deberán proveer datos demográficos y clínicos de cada participante: de exploración física, resultados de estudios complementarios como analíticas sanguíneas, estudios electrofisiológicos, resonancia magnética muscular y biopsia muscular si los hubiera. Estos datos se ingresarán en una plataforma en línea (PhenoStore) que utiliza la Ontología del Fenotipo Humano (HPO) para estandarizar la información clínica. Así mismo, en la medida de los posible, también se tomarán muestras de padres y familiares, tanto afectos como sanos. El equipo de coordinación de Newcastle se encargará de gestionar y costear el envío de las muestras desde los centros referentes a Newcastle.
Estrategia de análisis:
- Cribado inicial:
Dependiendo de la sospecha clínica indicada por el medico referente, las muestras se someterán a una serie de análisis genéticos previos a la secuenciación de exoma. Estos incluyen: MLPA para distrofia muscular de Duchenne/Becker, Atrofia Muscular Espinal (AME) y neuropatías del tipo CMT1A, y análisis de mutaciones recurrentes puntuales para aquellos con sospecha de Miastenia congénita y enfermedad mitocondrial. Se estima que este análisis permitirá diagnosticar un 20-30% de los casos analizados. Los restantes, se someterán a secuenciación de exoma. - Secuenciación de exoma probando:
Las muestras de ADN de los casos índices y sus familiares afectos se enviarán al Centro Nacional de Análisis Genómico (CNAG) en Barcelona, España, donde se llevará a cabo la secuenciación del exoma completo y el procesamiento bioinformático inicial. Los datos procesados se subirán a la Plataforma de Análisis Genoma-Fenoma – GPAP de RD-Connect, donde se realizará el filtrado de variantes siguiendo estrategias estándares para enfermedades raras (por ejemplo, frecuencia poblacional y predictores de efecto de variante) para enfocarse en aquellas con mayor relevancia clínica. Las variantes identificadas serán clasificadas siguiendo las reglas del ACMG (American College of Medical Genetics and Genomics).
Luego, el análisis de datos y se hará de forma escalonada. En la primera etapa, se realizará la Secuenciación del Exoma Completo (WES) de los casos índice y familiares afectos, y se utilizará un panel in-silico que abarca más de 600 genes asociados con enfermedades neuromusculares (https://pubmed.ncbi.nlm.nih.gov/36697115/) Este enfoque inicial se espera que resuelva entre el 40% y el 60% de los casos. - Investigación Exhaustiva:
En una etapa posterior , los casos aun no resueltos serán examinados con mayor profundidad, guiados por el fenotipo y la historia clínica de cada paciente: se analizarán genes afuera del panel inicial, y/o se procederá a secuenciar a los padres (trio exoma). Este análisis en trío o en familia nos ayudará a esclarecer diagnósticos complejos, y dará posibilidad de identificar nuevos genes aun no asociados con enfermedades neuromusculares. - Discusión de casos:
Una vez realizada la primera ronda de análisis, se llevarán a cabo discusiones de casos, entre los colaboradores locales y el grupo coordinador, con el objetivo de evaluar la relevancia clínica de las variantes encontradas y planificar el manejo adecuado para cada paciente. - Almacenamiento y Acceso a Datos:
Todos los datos genómicos se almacenarán en la plataforma de acceso restringido GPAP, facilitando el análisis colaborativo y la revisión crítica de los hallazgos clínicos y genéticos.
Publicaciones:
El proyecto Latin-SEQ valora la contribución y el esfuerzo de todos y cada uno de los centros referentes y colaboradores. Para garantizar una representación justa y colaborativa, cualquier publicación de alcance general realizada por el equipo coordinador en Newcastle incluirá como coautor a cada uno de los miembros del Consorcio Latin-SEQ que hayan aportado muestras al estudio. Esta inclusión asegura el reconocimiento adecuado de todos los participantes y promueve la visibilidad del trabajo colectivo en la comunidad científica. Así mismo, cada centro tendrá plena libertad de liderar los trabajos y/o presentaciones de casos locales e individuales. Esta política de publicación fomenta la transparencia, la colaboración internacional y el avance científico, permitiendo que los hallazgos y los conocimientos generados a través del proyecto Latin-SEQ sean compartidos ampliamente, beneficiando a la comunidad médica y científica y a los pacientes con enfermedades neuromusculares.
Qué es Latin-SEQ:
Latin-SEQ es un proyecto de investigación cuyo objetivo principal es proporcionar diagnóstico genético a personas afectadas por enfermedades neuromusculares (ENM) hereditarias residentes en países de Latinoamérica (LATAM). Con este fin, utilizamos una serie de técnicas genéticas, fundamentalmente «Next Generation Sequencing» (NGS) pero también secuenciación tradicional usando Sanger y técnicas de MLPA. Se trata de un estudio internacional y multicéntrico en el que participan más de 60 centros hospitalarios en 18 países, coordinado por el John Walton Muscular Dystrophy Research Center (JWMDRC) en la Universidad de Newcastle, Reino Unido.
Nuestro compromiso es acelerar el proceso de diagnóstico genético de estos pacientes y expandir el conocimiento sobre las causas y la prevalencia de las enfermedades neuromusculares en diferentes poblaciones LATAM. Obtener un diagnóstico precoz ofrece a los pacientes una multitud de beneficios: acceso a un plan de seguimiento y terapia personalizada, expectativas claras sobre la progresión de su enfermedad, posibilidad de obtener consejo genético para los familiares, posibilidad de inclusión en registros o asociaciones de pacientes y, participación en ensayos clínicos si los hubiese.
Latin-SEQ fomenta la creación de una sólida red de unidades hospitalarias y centros clínicos con experiencia en enfermedades neuromusculares que siguen a pacientes adultos y pediátricos abarcando toda LATAM.
Latin-SEQ también incluye espacios virtuales para la formación especializada, el debate conjunto de casos clínicos y la colaboración en proyectos de investigación reuniendo a neurólogos, neuropediatras, neurofisiólogos, genetistas, fisioterapeutas y otros profesionales de la salud que estén involucrados en el diagnóstico y seguimiento de pacientes con enfermedades neuromusculares.
Objetivos
Qué es secuenciado de exomas:
La secuenciación es una técnica molecular que permite analizar el ADN de una persona para “leer” su código genético. Anteriormente, se utilizaban métodos como la secuenciación de Sanger, que, aunque preciso, era más lento, ya que solo permite secuenciar una única región del ADN (gen) a la vez.
Hoy en día, utilizamos técnicas más avanzadas como la Secuenciación de Nueva Generación (NGS). Esta nueva tecnología es más rápida y permite secuenciar grandes regiones de ADN simultáneamente y en poco tiempo, lo cual facilita y acelera el diagnóstico genético. En el proyecto Latin-SEQ, usamos Secuenciación de Exoma Completo (WES por sus siglas en inglés), a modo de obtener información de todas las regiones codificantes, es decir aquellas que contienen las instrucciones para producir todas las proteínas. Al comparar los exomas de un individuo afecto, con una secuencia de referencia, podemos identificar variantes genéticas que puedan ser la causa de estas enfermedades neuromusculares. Gracias a esta tecnología, podemos ofrecer un diagnóstico más rápido y preciso, mejorando así la atención y la calidad de vida de los pacientes.
Criterios de inclusión y exclusión:
Pueden ser incluidos en el proyecto Latin-SEQ todos aquellos pacientes con sospecha de enfermedad neuromuscular de origen genético aun sin diagnóstico. No hay restricción de sexo ni de edad (pacientes adultos como pediátricos), ni tampoco respecto al tipo de enfermedad neuromuscular que presenten (tanto músculo y placa como nervio); solo quedaran excluidas las enfermedades adquiridas o aquellas que no puedan ser diagnosticadas mediante el análisis de exoma (DM1/2 y FSHD). El estudio priorizará el análisis de familias con múltiples miembros afectados y pacientes de minorías étnicas.
Reclutamiento, toma de muestras y fenotipado:
Los profesionales sanitarios de cada centro participante seleccionarán sus pacientes y se asegurarán de que los participantes firmen el consentimiento informado. Además, se encargarán de tomar las muestras biológicas (ADN, sangre, saliva o gotas de sangre seca en función de sus posibilidades). Estas serán enviadas y almacenadas en el biobanco del “JWMDRC Biobank” en Newcastle, hasta su análisis. Además, deberán proveer datos demográficos y clínicos de cada participante: de exploración física, resultados de estudios complementarios como analíticas sanguíneas, estudios electrofisiológicos, resonancia magnética muscular y biopsia muscular si los hubiera. Estos datos se ingresarán en una plataforma en línea (PhenoStore) que utiliza la Ontología del Fenotipo Humano (HPO) para estandarizar la información clínica. Así mismo, en la medida de los posible, también se tomarán muestras de padres y familiares, tanto afectos como sanos. El equipo de coordinación de Newcastle se encargará de gestionar y costear el envío de las muestras desde los centros referentes a Newcastle.
Estrategia de análisis:
- Cribado inicial:
Dependiendo de la sospecha clínica indicada por el medico referente, las muestras se someterán a una serie de análisis genéticos previos a la secuenciación de exoma. Estos incluyen: MLPA para distrofia muscular de Duchenne/Becker, Atrofia Muscular Espinal (AME) y neuropatías del tipo CMT1A, y análisis de mutaciones recurrentes puntuales para aquellos con sospecha de Miastenia congénita y enfermedad mitocondrial. Se estima que este análisis permitirá diagnosticar un 20-30% de los casos analizados. Los restantes, se someterán a secuenciación de exoma. - Secuenciación de exoma probando:
Las muestras de ADN de los casos índices y sus familiares afectos se enviarán al Centro Nacional de Análisis Genómico (CNAG) en Barcelona, España, donde se llevará a cabo la secuenciación del exoma completo y el procesamiento bioinformático inicial. Los datos procesados se subirán a la Plataforma de Análisis Genoma-Fenoma – GPAP de RD-Connect, donde se realizará el filtrado de variantes siguiendo estrategias estándares para enfermedades raras (por ejemplo, frecuencia poblacional y predictores de efecto de variante) para enfocarse en aquellas con mayor relevancia clínica. Las variantes identificadas serán clasificadas siguiendo las reglas del ACMG (American College of Medical Genetics and Genomics).
Luego, el análisis de datos y se hará de forma escalonada. En la primera etapa, se realizará la Secuenciación del Exoma Completo (WES) de los casos índice y familiares afectos, y se utilizará un panel in-silico que abarca más de 600 genes asociados con enfermedades neuromusculares (https://pubmed.ncbi.nlm.nih.gov/36697115/) Este enfoque inicial se espera que resuelva entre el 40% y el 60% de los casos. - Investigación Exhaustiva:
En una etapa posterior , los casos aun no resueltos serán examinados con mayor profundidad, guiados por el fenotipo y la historia clínica de cada paciente: se analizarán genes afuera del panel inicial, y/o se procederá a secuenciar a los padres (trio exoma). Este análisis en trío o en familia nos ayudará a esclarecer diagnósticos complejos, y dará posibilidad de identificar nuevos genes aun no asociados con enfermedades neuromusculares. - Discusión de casos:
Una vez realizada la primera ronda de análisis, se llevarán a cabo discusiones de casos, entre los colaboradores locales y el grupo coordinador, con el objetivo de evaluar la relevancia clínica de las variantes encontradas y planificar el manejo adecuado para cada paciente. - Almacenamiento y Acceso a Datos:
Todos los datos genómicos se almacenarán en la plataforma de acceso restringido GPAP, facilitando el análisis colaborativo y la revisión crítica de los hallazgos clínicos y genéticos.
Publicaciones:
El proyecto Latin-SEQ valora la contribución y el esfuerzo de todos y cada uno de los centros referentes y colaboradores. Para garantizar una representación justa y colaborativa, cualquier publicación de alcance general realizada por el equipo coordinador en Newcastle incluirá como coautor a cada uno de los miembros del Consorcio Latin-SEQ que hayan aportado muestras al estudio. Esta inclusión asegura el reconocimiento adecuado de todos los participantes y promueve la visibilidad del trabajo colectivo en la comunidad científica. Así mismo, cada centro tendrá plena libertad de liderar los trabajos y/o presentaciones de casos locales e individuales. Esta política de publicación fomenta la transparencia, la colaboración internacional y el avance científico, permitiendo que los hallazgos y los conocimientos generados a través del proyecto Latin-SEQ sean compartidos ampliamente, beneficiando a la comunidad médica y científica y a los pacientes con enfermedades neuromusculares.